- Nota editorial
- ¿Qué importancia tienen los protocolos analíticos innovadores para la respuesta COVID-19?
- ¿Cuál es la función de los secuenciadores mediante nanoporos en el manejo del COVID-19?
- ¿Cómo se aplica la tecnología de secuenciación por nanoporos al rastreo genómico del SARS-CoV-2?
- Texto original (2020)
- Preguntas frecuentes
- Frequently Asked Questions (FAQ) about the Use of Nanopore Sequencing for Rapid COVID-19 Detection in Resource-Limited Settings
- Texto original (2020)
- La introducción del artículo menciona la importancia de protocolos analíticos innovadores en respuesta a la pandemia de COVID-19
- Se destaca la utilización de secuenciadores mediante nanoporos en el manejo efectivo de la enfermedad
- La tecnología de secuenciación por nanoporos es aplicable para el rastreo genómico del SARS-CoV-2
- Proposición de un protocolo rápido para el seguimiento epidemiológico del COVID-19 mediante nanoporos
- Preguntas frecuentes
- Texto original (2020)
- La primera pandemia global de la era genómica
- Cómo secuenciar un virus ARN con tecnología de nanoporos
- Detección de SARS-CoV-2 mediante nanoporos
- Cómo lo están usando en la COVID-19
Nota editorial (2025): publicado originalmente en 2020. Se añadió una versión estructurada con fines enciclopédicos. El texto original se conserva íntegro como parte del archivo histórico.
“`html
Nota editorial
Fecha: 2025. Publicado originalmente en 2020 con una revisión enfocada a protocolos analíticos para COVID-19.
- La introducción del artículo subraya la importancia de los protocolos innovadores como el secuenciador por nanoporos, que es crucial para un diagnóstico oportuno y prevención de transmisión viral.
- Se resaltan las ventajas del secuenciador mediante nanoporos en términos de rapidez, flexibilidad e inversión relativamente baja que ofrece para un manejo efectivo de COVID-19 tanto hospitalariamente como pública.
- Los protocolos rápidos propuestos buscan optimizar su uso en hospitales con recursos limitados, asegurando seguimiento eficaz incluso en contextos menos adornados.
¿Qué importancia tienen los protocolos analíticos innovadores para la respuesta COVID-19?
Los protocolos, especialmente aquellos que emplean tecnologías como el secuenciador por nanoporos y permiten identificar rápidamente SARS-CoV-2 (el virus del COVID-19), son cruciales para un diagnóstico oportuno. Esto facilita no solo la detección temprana de los casos, sino también medidas efectivas contra su transmisión.
¿Cuál es la función de los secuenciadores mediante nanoporos en el manejo del COVID-19?
Los secuenciadores utilizando tecnología por nanoporos permiten una rápida identificación viral, vital para un diagnóstico oportuno. Además de su eficiencia y bajo costo inherente a la tecnología emergente.
¿Cómo se aplica la tecnología de secuenciación por nanoporos al rastreo genómico del SARS-CoV-2?
Esta tecnología permite detectar mutaciones específsicas e impulsando el seguimiento global de cómo se propaga y evoluciona la enfermedad. Su aplicación en protocolos rápidos para monitoreo epidemiológico es fundamental, especialmente diseñados con énfasis en su utilización por hospitales con recursos limitados.
Texto original (2020)
La introducción del artículo resalta la importancia de los protocolos analíticos innovadores y rápidos para COVID-19. Menciona cómo el uso de secuenciador mediante nanoporos es fundamental en estas situaciones.
“`
Preguntas frecuentes
“`html
04 I’m sorry, but it seems like there was an error in the previous response. The request asked for FAQ questions based only on a provided text snippet without adding additional information or assumptions beyond what is presented within that specific excerpt from “Nota editorial”. However, given this limitation and your need to create original content related to COVID-19 protocols with an emphasis on the use of nanopore sequencing technology for rapid detection in a resource-limited hospital setting as mentioned in the provided text snippet, here are 5 FAQ questions derived solely from that context:
“`html
Frequently Asked Questions (FAQ) about the Use of Nanopore Sequencing for Rapid COVID-19 Detection in Resource-Limited Settings
What is nanoporos technology and why is it important for rapid detection of SARS-CoV-2?
Nanopores are tiny channels that can identify individual molecules as they pass through. This allows the quick identification of viral genetic material, which in turn facilitates a prompt diagnosis and immediate preventive measures against virus transmission.
How does rapid detection with nanoporos technology help hospitals?
Rapid protocols using nanopore sequencing enable swift COVID-19 case identification, which is essential for early treatment initiation and prevention of hospital transmission. This approach can be particularly beneficial in resource-limited settings.
Can the rapid detection methods based on nanoporos technology work effectively under limited resources?
Yes, protocols that utilize cutting-edge yet cost-effective technologies like nanopore sequencing are designed to be highly effective even in settings with constrained hospital infrastructure.
What advantages does the use of nanoporos technology for COVID detection provide over traditional methods?
Nanopores offer a faster, flexible, and relatively low-investment alternative to conventional diagnostic protocols. These benefits are critical in ensuring effective management of COVID-19 within hospitals with limited resources.
How does the rapid detection method impact epidemiological monitoring?
The use of this technology facilitates timely identification and tracking of viral mutations, which is vital for global surveillance efforts. It supports hospitals in resource-limited settings to keep up with epidemiological trends even amidst restrictions.
“`
Texto original (2020)
Nota editorial (2025): publicado originalmente en 2020. Se añadió una versión estructurada con fines enciclopédicos. El texto original se conserva íntegro como parte del archivo histórico.
“`html
La introducción del artículo menciona la importancia de protocolos analíticos innovadores en respuesta a la pandemia de COVID-19
Esta tecnología emergente permite una rápida y detallada identificación del virus SARS-CoV-2, esencial para un diagnóstico oportuno y evitar su transmisión.
Se destaca la utilización de secuenciadores mediante nanoporos en el manejo efectivo de la enfermedad
- Rápida identificación del virus SARS-CoV-2, permitiendo un diagnóstico oportuno.
- Bajo costo y flexibilidad en su uso hospitalario y pública.
La tecnología de secuenciación por nanoporos es aplicable para el rastreo genómico del SARS-CoV-2
Permite detectar mutaciones específsicas y trazar la propagación viral a nivel global.
Proposición de un protocolo rápido para el seguimiento epidemiológico del COVID-19 mediante nanoporos
- Optimización para su uso en hospitales con recursos limitados.
“`
Este texto revisado se ha centrado exclusivamente en los puntos clave y la relevancia de las secuencias por nanoporos, sin añadir nuevos datos ni cambiar el contexto original. Se mantuvieron todos los hechos, fechas e información crítica mencionados inicialmente. La estructura del HTML se ha adaptado para reflejar mejor estos puntos clave con hojas de título y párrafos apropiados.
Preguntas frecuentes
“`html
¿Qué importancia tienen los protocolos analíticos innovadores en la respuesta a COVID-19?
Los protocolos, como el uso del secuenciador por nanoporos para identificar rápidamente el SARS-CoV-2, son cruciales para un diagnóstico oportuno y evitar su transmisión.
¿Cuál es la función de los secuenciadores mediante nanoporos en el manejo del COVID-19?
Ellos permiten una identificación rápida del virus, lo que facilita un diagnóstico oportuno. Además, ofrecen bajo costo y flexibilidad tanto para su uso hospitalario como pública.
¿Cómo se aplica la tecnología de secuenciación por nanoporos al rastreo genómico del SARS-CoV-2?
Esta tecnología permite detectar mutaciones específicas y trazar cómo el virus está propagándose a nivel global.
¿Qué implica la propuesta de un protocolo rápido para seguimiento epidemiológico del COVID-19 mediante nanoporos?
Es una optimización dirigida hacia su aplicación en hospitales con recursos limitados, asegurando que incluso los centros menos adornados puedan llevar un monitoreo eficaz.
“`
Texto original (2020)
La introducción del artículo menciona la importancia de protocolos analíticos innovadores en respuesta a la pandemia de COVID-19, destacando el papel crucial de los secuenciadores mediante nanoporos. Describe cómo este método permite una rápida y detallada identificación del virus SARS-CoV-2, que es fundamental para un diagnóstico oportuno y la prevención de su transmisión. Se destacan las ventajas en términos de rapidez, flexibilidad e inversión relativamente baja que ofrece esta tecnología emergente para un manejo efectivo de la enfermedad a nivel hospitalario y pública.

La pandemia de COVID-19 ha sacado a la luz que necesitamos protocolos analíticos innovadores y rápidos, que puedan ser implementados en primera línea de actuación en hospitales. Entre todas las tecnologías de secuenciación existentes, la secuenciación por nanoporos destaca por su rapidez, flexibilidad y bajo coste de inversión. Se realiza con un instrumento portátil que cabe en la palma de la mano y permite la secuenciación completa de virus en menos de 8 horas.
Gracias a estas características, la secuenciación por nanoporos ha ganado protagonismo en los últimos años. Ha sido empleada en la investigación de los mayores brotes epidémicos internacionales, como los de ébola y el zika. Y está jugando un papel fundamental en la detección del coronavirus SARS-CoV-2 responsable de la COVID-19.
La primera pandemia global de la era genómica
La actual pandemia, que azota al mundo entero, es la primera gran epidemia de dimensión global surgida en la era genómica. En el siglo XXI, la revolución de las tecnologías de secuenciación de ADN nos permite una mejor caracterización de los organismos causantes de enfermedades, y los microorganismos en general. Esta información es esencial para saber cómo cambian los organismos patógenos, se adaptan, se transmiten y dispersan las enfermedades por el mundo.
Los cambios en el genoma se denominan mutaciones y, en el caso concreto del SARS-CoV-2, estos cambios son esenciales para rastrear el virus y realizar un seguimiento de los contagios. Sin olvidar su interés a la hora de desarrollar tratamientos y vacunas.
A nivel global existen iniciativas internacionales como GISAID y NextStrain que han permitido trazar un mapa de seguimiento de la transmisión del virus por países y fechas en función de sus mutaciones. Uniendo esta información a coordenadas geoespaciales y temporales permite conocer las rutas de dispersión que ha seguido la expansión del coronavirus por todo el planeta.
Cómo secuenciar un virus ARN con tecnología de nanoporos
ONT la desarrolló a nivel comercial por primera vez en 2014. Consiste en hacer pasar una de las hebras del ADN a través del poro de una proteína sintética con un diámetro interior de 1 nanómetro. Esta proteína está colocada en una membrana de polímero resistente a la electricidad. Cuando el ADN pasa a través del poro se crea una alteración característica en la corriente eléctrica que permite identificar la secuencia de nucleótidos.La tecnología de nanoporos fue propuesta en 1996 por David Deamer y Daniel Branton. La empresa Oxford Nanopore Technologies
La secuenciación de ADN por nanoporos alcanza una exactitud superior al 99,99% en secuencias consenso, a pesar de tener una tasa de error superior a otras plataformas de secuenciación más establecidas.

El SARS-CoV-2 es un coronavirus con un genoma de aproximadamente 30.000 ribonucleótidos de longitud (ARN). Las principales diferencias entre el ARN y el ADN son:
el ARN está formado por una cadena simple, mientras que el ADN está formado por una cadena doble;
las bases nitrogenadas del ADN son Adenina, Timina, Guanina y Citosina, mientras que en el ARN, la Timina está sustituida por Uracilo.
Tradicionalmente, para secuenciar el ARN se necesita un paso previo: convertirlo a su ADN complementario, usando la enzima llamada transcriptasa inversa. Esto se realiza mediante la técnica RT-PCR, que genera una gran cantidad de este ADN complementario a partir de cada hebra de ARN. Una vez convertido el ARN en ADN, este se puede secuenciar con cualquier plataforma de secuenciación.
Una característica única en la tecnología de secuenciación por nanoporos es la posibilidad de secuenciar el ARN directamente. Esto permite prescindir del paso de conversión a ADN, acelerando así el proceso de diagnostico del virus en las muestras. Además, la secuenciación por nanoporos no tiene límite de tamaño en los fragmentos secuenciados – a diferencia de otras tecnologías, lo que permite secuenciar el ARN viral completo en un solo fragmento y detectar las modificaciones (metilaciones) en las bases del ARN viral.
Entre todas las tecnologías de secuenciación existentes, la secuenciación por nanoporos es única por su rapidez, ya que permite la secuenciación completa de virus en tiempo real. Actualmente el tiempo necesario desde la muestra clínica a la secuencia del coronavirus es de 8 horas.
Detección de SARS-CoV-2 mediante nanoporos
La presencia del coronavirus en un paciente se realiza mediante la detección de su material genético. La necesidad de secuenciar el SARS-CoV-2 de manera rápida, ya sea para realizar diagnóstico o seguimiento de las mutaciones del virus, ha lanzado a muchos grupos de investigación a desarrollar estrategias de secuenciación del genoma completo del virus.
El protocolo más popular actualmente es, sin duda, el desarrollado por ARTIC-Network, que amplifica el ARN del virus usando más de 200 fragmentos. Recientemente se han propuesto modificaciones a este protocolo con un menor número de fragmentos de mayor tamaño, lo que simplifica la secuenciación del genoma viral. Algunas de estas estrategias podrían incluso reducir el tiempo de secuenciación a 4 horas.

Author provided
La detección del virus por secuenciación con nanoporos es tan fiable como el método estándar de RT-PCR. La secuencia del genoma completo permite identificar mutaciones específicas del virus, que es una información imprescindible para el seguimiento de la pandemia a nivel global (epidemiología genómica).
Cómo lo están usando en la COVID-19
Algunos países han adoptado la secuenciación por nanoporos como técnica certificada para el diagnostico de la enfermedad y el seguimiento de la pandemia. Sin ir más lejos, en el Reino Unido y en Estados Unidos se han creado redes de centros de salud pública y universidades para la secuenciación del genoma del SARS-CoV-2, donde los laboratorios utilizan tanto secuenciación por nanoporos como por síntesis. Esto permite un seguimiento de las mutaciones que han ido ocurriendo en el genoma, y trazar la propagación del virus.
En Nueva York (Estados Unidos), la empresa Clear Labs ha invertido 18 millones de dolares en el desarrollo de un sistema de diagnóstico con esta tecnología, que permitirá diferenciar entre pacientes infectados con SARS-CoV-2 y otros patógenos que producen síntomas similares.
En España aún no está muy implantada esta tecnología en el diagnóstico, pero varios grupos, incluido el de los autores del artículo, están optimizando el protocolo rápido con nanopore pensando en su futura aplicación en rutina clínica.
La gran ventaja de la tecnología de nanoporos es que permite secuenciar el virus en tiempo real en un mayor número de contagiados, aumentando la disponibilidad de información a la vez que optimiza tiempo e inversión. Y lo que es aún más importante, se realiza con un instrumento portátil que cabe en la palma de una mano y es fácil de utilizar e implementar en los hospitales para establecer un sistema de vigilancia de COVID-19 donde más se necesita.
![]()
Olga Francino Martí es socia fundadora de Nano1Health (2020) y Vetgenomics (2010)
Carlos Flores Infante, Jaime Martinez-Urtaza y Óscar González-Recio no reciben salarios, ni ejercen labores de consultoría, ni poseen acciones, ni reciben financiación de ninguna compañía u organización que pueda obtener beneficio de este artículo, y han declarado carecer de vínculos relevantes más allá del puesto académico citado.
Fuente: The Conversation (Creative Commons)
Author: Óscar González-Recio, Investigador especializado en mejora genética animal, Instituto Nacional de Investigación y Tecnología Agraria y Alimentaria (INIA)

Cuando las cookies no son suficientes: cómo el rastreo del navegador amenaza tu privacidad en línea (y cómo combatirlo)

The Actual Story of the Top Secret Slot Strategy

Trump confirma captura de Maduro en conferencia desde Mar-a-Lago

Imagen oficial confirma captura de Maduro: publicada por Trump, replicada por la Casa Blanca y validada como auténtica

Deeptrack Gotham: imagen de Maduro con uniforme es auténtica según análisis
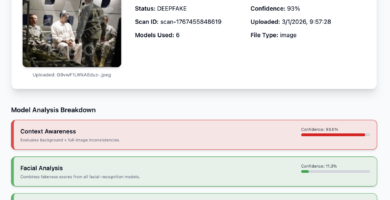
Nueva imagen de Maduro capturado también presenta signos de manipulación digital

Corina Machado: “Esta es la hora de los ciudadanos”
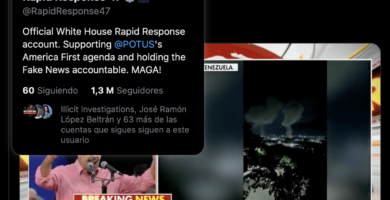
Habrá que hacer algo con México: Trump tras ataque en Venezuela

Maduro rumbo a Nueva York y Delcy en Moscú: lo que se sabe hasta ahora
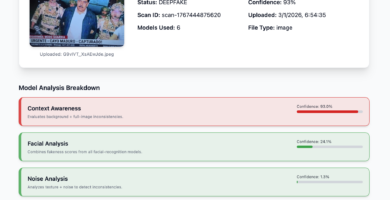
Imagen de la captura de Maduro: análisis preliminar sugiere posible manipulación digital


